搜索

在预测生物学中,基因合成网络受到非常多的关注,尤其是由少数几个基因或物质组成的基本的单元模块(motif)。不同的模块因不同的结构拥有不同的表现形式,进而实现不同的功能。本篇文章介绍两种经典的基因调控模型,toggle switch[1]和repressilator[2],并观察它们结合之后的行为[3]。
1. toggle switch
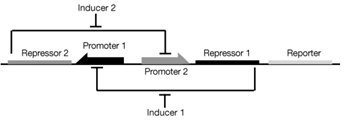
拨动开关(toggle switch)由两个抑制子(Repressor)和两个启动子(Promoter)组成,其中每个启动子都被另一个启动子转录的阻遏物抑制,T型箭头即表示抑制作用,这个符号也是一般基因表达模型中的规范符号。
这个简单调控元件只有两个基本组成部分,并且没有显式的自我调控,然而一对相互抑制的调控反馈(负反馈循环)已经可以使系统产生一种有趣的现象——双稳态。2000年发表在Nature上的文章[1]即在大肠杆菌中构建了这样的基因拨动开关,并成功观察到了相关的现象。
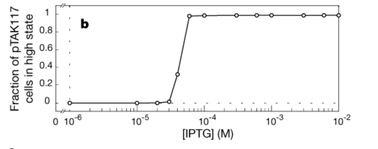
如图,质粒有两种状态:低状态或者高状态,随着IPTG浓度的增加,在浓度到达~区间的时候,pTAK117翻转质粒显示出到高状态的准间断跳跃。这种状态的突然切换就是所谓的双稳态显现,其与数学中的分岔理论有关。
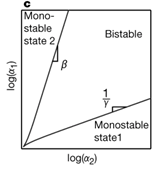
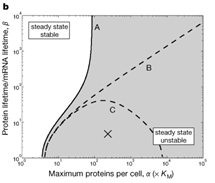
如图所示,参数与基因表达的曲线实际蕴含着鞍-结分岔,两个鞍形节点分叉确定了两个稳定解的稳定范围(黑色实线)。中间信号所对应的系统具有双稳态,其中两个稳定解都由不稳定的平衡态(虚线)分开。粉红色箭头对应着达到稳态动态过程。
该模型的无量纲化数学形式如下:
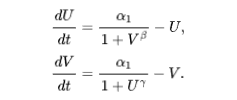
其中、表示两种阻遏物的浓度,、表示合成的有效速率,、是从基因表达的生物化学速率方程中得到的(整数)。
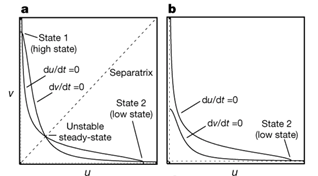
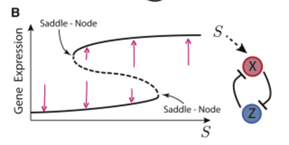
双稳态是由两零线(即变化速率为零的线)的交点产生的,当两阻遏物变化速率都为零时,系统处于平衡状态。图a中上下两点分别为高状态和低状态,是稳定的,而中间的为不稳定的平衡态,图b则没有双稳态产生,这与系统中的参数有关。双稳态的产生条件,由模型进行计算,可以完全给出。
2. repressilator
第二个经典的组件,由三个依次抑制的基因构成,它们所形成的负反馈回路,保证了稳定振荡产生的可能。
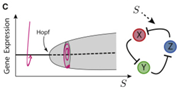
在由外部信号S控制的参数变化下,基因表达经历了一个Hopf分岔,系统产生了稳定的极限环,从而产生了稳定的振荡行为,这种周期性的稳定振荡与生物体内的节律行为有关。下图中,稳定的螺旋(阻尼振荡)(黑色实线)变得不稳定(虚线),从而导致稳定的振荡(阴影区域)产生。系统经历分叉之后,稳定振荡的振幅(由细实线界定)随信号而增加。粉色的线表示了给定参数下,基因表达可能的动态变化。
该模型的无量纲化数学表达式由六个耦合的一阶微分方程组成:
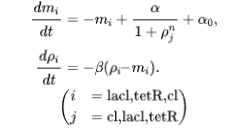
其中,分别表示为mRNA浓度与蛋白质浓度,不同的参数、决定了方程系统的稳定区域。
图中c为稳定的振荡行为,而d为Hopf分岔之前的阻尼震荡。
3. AC-DC
十八年之后,发表在Cell Systems上的文章[3]将这两种基本组件结合,并命名为AC-DC(交流-直流)电路。AC-DC电路的名称类似于AC和DC,因为它能够产生振荡(AC)和多稳态(DC)行为。
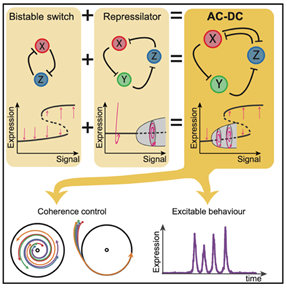
研究发现,由两种motif产生的振荡和稳定表达式可以在参数空间的较大区域中共存,也就是说,这是一种新的小规模多功能基因网络。AC-DC电路最初是在脊椎动物神经管中祖细胞的图案中观察到的[4]。
如前面的模型一样,我们用无量纲形式的方程组来刻画这个网络,,,分别表示三个基因的表达程度(蛋白质浓度):
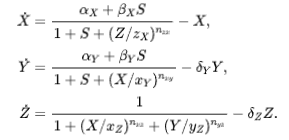
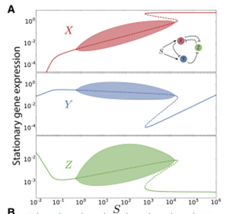
而此时随信号变化的基因表达结合了之前性质,在某些参数下,初始时刻不同的基因浓度可能导致最终结果的不同——稳定周期振荡或者稳定的平衡态。
上图中,粗实线表示稳定状态,虚线表示不稳定状态。阴影区域显示了振荡状态的表达范围,该范围由细实线界定。
更重要的是,AC-DC电路可以通过两种不同的机制来开启振荡,将相干性引入到振荡器的集合中。
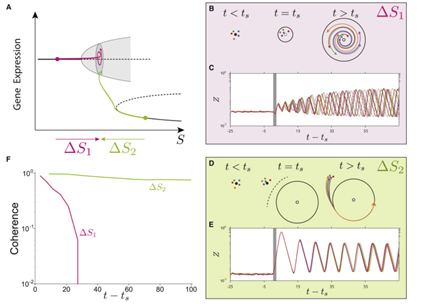
当信号从高和低两个不同方向向中间改变至同一个值的时候,会发现系统产生两个不同的振荡,大振幅与小振幅,这种差异导致振荡状态的不同动力学瞬态。在第一种情况下,从一个低值增加信号会引起细胞群的异步振荡。初始阶段的微小差异会随着时间的推移而放大。相比之下,第二种情况是,从高电平减少信号会在噪声面前引起相干振荡(由鞍-结分岔产生)。
总结
这里,我们只简要介绍了两种基本的基因网络和其结合之后的网络,更多的性质和应用都没有涉及。我们的关注点主要在于基本网络模型对于预测生物学的指示作用,复杂的现象有可能是由于多个简单模型耦合而产生的。
文:仲昭月
【参考文献】
Gardner, T.S., C.R. Cantor, and J.J. Collins, Construction of a genetic toggle switch in Escherichia coli. Nature, 2000. 403(6767): p. 339-342.
Elowitz, M.B. and S. Leibler, A synthetic oscillatory network of transcriptional regulators. Nature, 2000. 403(6767): p. 335-338.
Perez-Carrasco, R.et al, Combining a toggle switch and a repressilator within the AC-DC circuit generates distinct dynamical behaviors. Cell Systems, 2018. 6(4): p. 521-530.e3.
Balaskas, N., et al., Gene regulatory logic for reading the sonic hedgehog signaling gradient in the vertebrate neural tube. Cell, 2012. 148(1): p. 273-284.


 loading......
loading......